Les études faisant appel au diagnostic (sensu lato, c'est-à-dire y compris celles impliquant une détermination des espèces, des souches, des populations ou des isolats de bioagresseurs) génèrent un grand nombre de données et métadonnées... Qui, souvent, méritent d'être exploitées davantage qu'elles ne le sont.
Partager les données
Les données issues des diagnostics en santé végétale sont produites par une large diversité d'opérateurs publics, professionnels ou privés : observateurs au champ, laboratoires de diagnostic, laboratoires nationaux de référence (LNR), laboratoires de recherche, etc. La nature et le volume de ces données, qui peuvent paraître disparates, correspondent d'abord à l'usage pour lequel elles ont été acquises : surveillance sanitaire, confirmation d'identification, typage pour la certification du matériel végétal, etc. Ces jeux de données brutes sont donc le plus souvent conservés par les institutions qui les ont acquis, sans considération ou tentative de partage.
Or, il est imaginable que l'exploitation concertée de jeux de données acquis initialement dans des objectifs différents puisse permettre d'apporter des réponses à d'autres questions pertinentes, non envisagées lors de la collecte des données.
Un des groupes de travail du RMT VegDiag s'est donc intéressé à ce cas de figure, avec une triple démarche :
- réaliser une typologie des jeux de données disponibles et de leurs applications actuelles ;
- analyser de manière prospective sur quelques cas emblématiques de cette typologie les conditions et sorties potentielles, et la faisabilité d'une analyse conjointe de jeux de données distincts ;
- identifier les nouvelles questions générées par un partage des données effectif et les conditions de réussite.
Classer l'existant
Première typologie des données et usages
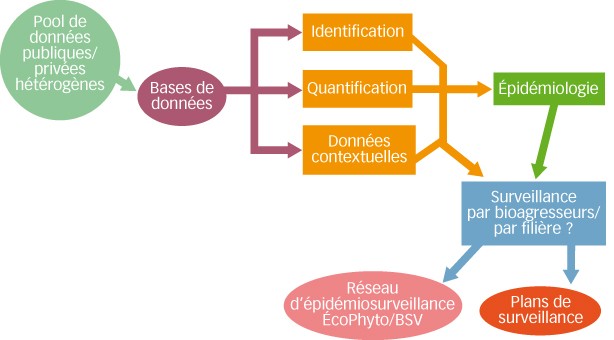
Une enquête a été réalisée auprès des membres du RMT afin d'identifier la nature des principales données existantes au sein de leur structure et d'en connaître les caractéristiques générales et usages (Figure 1).
Une dizaine de structures ont répondu à cette enquête, certaines ayant renseigné plusieurs jeux de données. Cet inventaire est donc loin d'être exhaustif, mais les réponses obtenues permettent toutefois de tenter une première typologie des données disponibles, selon trois grands critères : origine, mode de collecte et usages initiaux.
Nature de l'échantillonnage sous-jacent
Les données peuvent être issues d'échantillonnages « passifs » ou « actifs ». Les données issues d'échantillonnages passifs liés à la surveillance événementielle (remontées spontanées de signalements) concernent essentiellement des résultats d'analyse d'échantillons prélevés pour diagnostiquer les causes d'un symptôme observé.
Ce type d'échantillons est généralement associé à très peu d'informations contextuelles, ce qui limite les possibilités d'exploitation des données acquises pour des utilisations autres que la cartographie des occurrences. Par ailleurs, la représentativité de ce type d'échantillonnage n'est pas garantie : elle est très dépendante de l'effort d'échantillonnage consenti, et donc des observateurs, d'autant plus si la surveillance repose sur des signalements spontanés. Par contre, ces données sont souvent utiles pour l'identification de nouveaux bioagresseurs, ou le suivi de leur expansion territoriale initiale.
Les données issues d'échantillonnages actifs, conduits dans le cadre soit d'actions d'épidémiosurveillance (parcelles cultivées et/ou parcelles sentinelles ou témoins), soit de projets de R&D, sont collectées directement sur le terrain selon un protocole (planification de l'échantillonnage). Elles peuvent concerner l'identification de bioagresseurs, ou la description de certains de leurs traits particuliers (pouvoir pathogène, sensibilité aux xénobiotiques, gamme d'hôte...).
L'important pour l'exploitation de ces jeux de données est alors :
- que la nature et la forme des données à partager (effectif de la population, stades de collecte, type de mesure...) soit identifiées ;
- que les modes de prise d'échantillons soient comparables.
Méthodologies d'acquisition des données
Ces données peuvent être issues d'observations de terrain avec ou sans analyses de laboratoire associées, ou encore d'expérimentations (biotests).
Les observations de terrain sans analyse de laboratoire sont les diagnostics visuels sur des échantillons individuels, et les données d'observation de réseaux d'épidémiosurveillance (type BSV, Bulletin de santé du végétal).
Les observations de terrain confortées par une analyse de laboratoire portent sur des échantillons individuels prélevés pour diagnostic initial, ou bien des vérifications planifiées de symptômes observés au champ dans quelques régions ou même sur toute la France. Elles proviennent aussi de données issues des premiers diagnostics en santé végétale (type BSV à dire d'experts, observations, laboratoires...). Les techniques d'analyse sont souvent moléculaires mais peuvent aussi associer des analyses morphobiométriques. Ces méthodes peuvent être officielles ou bien internes. Les résultats de biotests sont généralement obtenus dans le cadre de projets de recherche/R&D : caractérisation de résistances aux maladies et aux ravageurs, caractérisation de populations de bioagresseurs pour leur pouvoir pathogène ou leur résistance à certains xénobiotiques, etc.
Usages initiaux
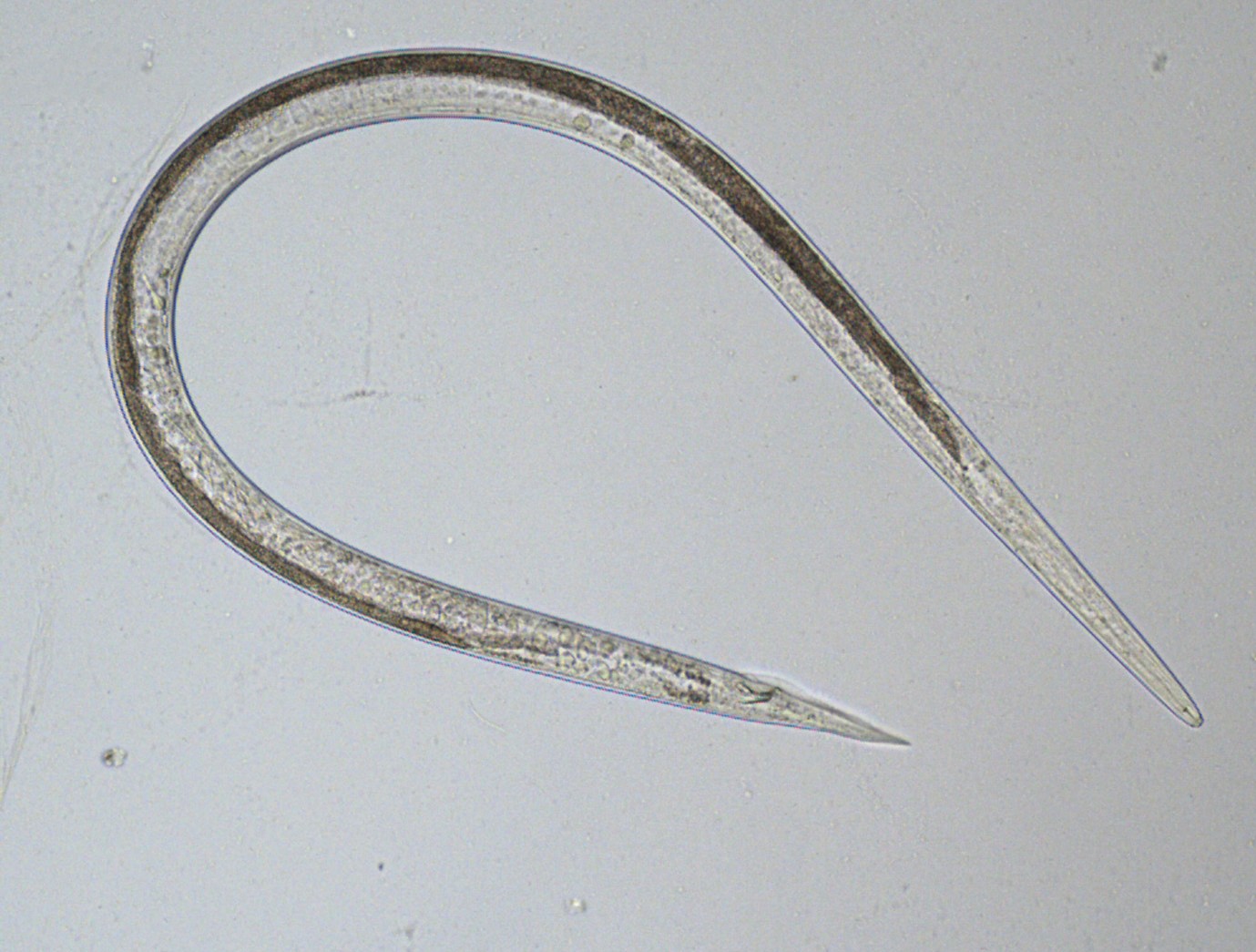
Les principales filières concernées sont les grandes cultures, les semences, les légumes et certaines cultures tropicales. Les retours d'enquête ont surtout porté sur des bactéries, des virus et des champignons, et dans une plus faible mesure des nématodes et des insectes. Les observations et analyses portent très généralement sur les végétaux eux-mêmes. Dans le cas des nématodes, en complément des observations directes sur plantes, les méthodes d'extraction portent aussi sur les racines, le bois dans le cas du nématode du pin, et aussi des semences (Ditylenchus, photo 1, sur luzerne).
Les principaux usages des données par les détenteurs ont trait :
- à la prévision des risques ;
- au suivi des pressions parasitaires ;
- à la cartographie des attaques et des émergences ;
- à la modélisation épidémiologique ; c'est dans ce dernier cas que les données sont le plus complètement renseignées.
Objectifs pour l'agrégation
Quatre grands types identifiés
On peut ainsi, par extrapolation, identifier quatre grands types d'objectifs pour la collecte puis l'agrégation des données de diagnostic :
- décrire un état sanitaire d'un lot de plantes/semences ou d'une parcelle intégrés dans un plan d'épidémiosurveillance réalisé vis-à-vis de différents organismes émergents ou invasifs (représentativité territoriale) ;
- comprendre (projet de R&D), suivre la phénologie d'un bioagresseur avec un nombre de données suffisantes pour envisager une modélisation de son développement et/ou de sa distribution ;
- prédire le risque, par des modèles validés par des données issues des réseaux d'observations et des données climatiques ;
- diffuser ; les données, généralement élaborées, sont destinées aux gestionnaires des risques pour les informer sur la prévision des risques sanitaires et/ou agronomiques.
En recoupant l'ensemble des informations ci-dessus, on obtient la typologie générale résumée dans le Tableau 1.
Parallèlement à cette première caractérisation des types de données existants, nous avons formalisé, sur la base d'une réflexion collective menée au sein du RMT VegDiag, les usages possibles, premiers ou dérivés, permis par ces jeux de données.
Étude de cas
Des études de cas ont été conduites pour identifier les questions nouvelles que l'agrégation de données actuellement dispersées permettrait d'adresser, les verrous à une telle agrégation et les conditions requises pour les lever. Elles correspondent à des situations différentes par la nature des jeux de données disponibles et par les questions qu'une analyse conjointe permettrait d'aborder. Voici l'exemple de deux de ces études.
Mildiou de la pomme de terre : quatre sources de données
L'objet de ce travail est d'utiliser conjointement des données de phénotypage au champ et au laboratoire, d'épidémiosurveillance et de génotypage pour retracer, puis prédire l'évolution des populations françaises de Phytophthora infestans (mildiou de la pomme de terre) et l'efficacité des résistances variétales.
Les bases de données mobilisables sont publiques (Inra), privées (Arvalis, BSV, Météo France) et issues d'actions à visée initiale différentes : recherche en biologie des populations, observations de surveillance biologique du territoire, enregistrement météorologiques et données prévisionnelles de climatologie.
Les questions posées sont :
- existe-t-il un lien, et si oui lequel, entre sévérité des épidémies et caractéristiques de pouvoir pathogène des populations de P. infestans ?
- le pouvoir pathogène détermine-t-il le pouvoir invasif des génotypes de P. infestans au sein des populations ?
- les clones actuellement présents ou émergents de P. infestans fragilisent-ils les résistances variétales introduites dans les variétés inscrites ?
Traiter ces questions requiert :
- pour la première, un accès à Histopest (base de données numérisée contenant l'ensemble des avertissements agricoles et des BSV) et aux données météo locales, mais aussi des méthodes d'explorations statistiques conjointes des données ; ceci peut impliquer un recodage de la sévérité des épidémies, une évaluation a posteriori du risque climatique via Mileos (un outil de prévision du risque), et des analyses descriptives et corrélatives multivariées ;
- pour les autres, essentiellement du temps pour mettre en forme (saisie de données historiques) et traiter des jeux de données existants en interne à l'Inra. Ces travaux sont en cours ou prévus dans le cadre de deux projets, IPMBlight 2.0 et PotStaR.
Nématodes : deux bases de données publiques issues de projets différents
Les jeux données disponibles sur les nématodes nuisibles aux cultures sont issus de deux projets CasDAR successifs (Tableau 2).
Le jeu de données 1 était issu d'un CasDAR grandes cultures : données issues des campagnes 2008, 2009 et 2010.
Les objectifs de cette étude étaient :
- de déterminer si les nématodes sont impliqués ou non dans les dégâts observés sur culture de colza au printemps ;
- d'identifier les principales espèces impliquées en lien avec l'origine géographique des échantillons analysés ;
- d'acquérir de nouvelles connaissances sur le cycle biologique de Meloidogyne artiellia (photo 2) sur colza dans les conditions climatiques du nord de la France (Buisson et al., 2014).
Le jeu de données 2 provient de l'enquête en culture pluriannuelle sur les nématodes Ditylenchus dipsaci et Ditylenchus gigas sur féverole des campagnes 2011, 2012 et 2013. L'objectif de cette enquête était d'acquérir des références françaises sur un bassin de production donné, sur lequel les nématodes objets du projet ont déjà été détectés, en réalisant des suivis de parcelles de multiplication de féverole afin :
- d'identifier des symptômes en parcelles, et établir des liens avec les symptômes visuels sur graines et les résultats de tests de détection au laboratoire ;
- d'analyser le rôle de facteurs agronomiques dans le processus d'apparition de nématodes dans les parcelles, et identifier les facteurs de risque (Vinsant-Le Lous et Janson, 2011, 2012 et 2013).
L'agrégation de ces jeux de données a permis de formuler quatre nouvelles questions :
- à l'échelle des zones de production communes aux deux jeux de données, est-ce que lorsque l'on observe M. artiellia en parcelles cultivées, on observe également D. dipsaci et/ou D. gigas ?
- existe-t-il des pratiques anthropiques favorisant/défavorisant l'installation ou l'exclusion de ces trois espèces de nématodes ?
- existe-t-il des conditions environnementales favorables ou défavorables à l'installation ou l'exclusion des trois nématodes ?
- quels sont les relations et les assemblages possibles entre ces trois espèces ?
Si les jeux de données ne permettent pas d'y répondre immédiatement, ils suscitent toutefois l'intérêt de la question.
Traiter ces questions requiert notamment :
- la reconstitution d'un jeu de données unique intégrant les variables contextuelles quantitatives et qualitatives communes aux deux jeux de données ;
- le recodage des dénombrements des différentes espèces de nématodes en classes communes ;
- tester l'effet du climat sur les six années considérées ;
- analyser les données de manières descriptives et statistiques ;
- interpréter les données en tenant compte du climat de l'année ;
- identifier les verrous.
Les campagnes de prélèvements ne se chevauchent pas : un effet climat significatif est possible. La puissance des analyses de données pourrait s'avérer relativement faible compte tenu du nombre de parcelles agricoles présentes dans les deux zones de production communes.
Quels enseignements ?
Le partage des données serait utile, mais sa réalisation est délicate
La quantité des données en santé végétale est considérable de par le nombre de filières concernées et le nombre de bioagresseurs. La constitution de bases de données structurées sur ce sujet en est en cours par tous les acteurs de la surveillance biologique du territoire (SBT). À titre d'exemple, de nombreuses données sont disponibles (base Epiphyt DGAL, Vigiculture Arvalis, VégéObs du réseau Fredon, données des coopératives et des sociétés privées (semences et phytosanitaires...).
Ces données sont stratégiques, que ce soit en appui aux politiques publiques (analyses de risque phytosanitaire, réglementation pour la gestion des émergences et des organismes nuisibles réglementés, surveillance...) ou pour le développement de méthodes intégrées de gestion de la santé des cultures.
C'est pourquoi la caractérisation de la qualité des données acquises, le partage de référentiels communs mais aussi le développement de méthodes analytiques et statistiques adéquates pour traiter des jeux de données hétérogènes sont des préalables indispensables à une valorisation optimale de ces données.
La problématique du partage des données et de leur traitement collaboratif reste délicate. En effet, indépendamment des problèmes réels d'interopérabilité des bases ou fichiers les contenant, et comme l'a montré le faible taux de retour à notre enquête, les structures acquérant ces données sont peu disposées à les présenter, voire à révéler ce qu'elles détiennent hors projets de recherche (c'est-à-dire tant qu'il n'existe pas de financement extérieur et de définition commune d'objectifs précis).
On ne peut ainsi que constater le peu d'échanges et de mobilisation partagée des données entre les acteurs de terrain et les laboratoires de recherche. Ceci explique peut-être, au moins en partie, la faible utilisation actuelle des données de diagnostic pour le développement ou la validation de modèles de prévision des risques phytosanitaires.
Perspectives : projets à co-construire, progrès méthodologiques à exploiter
Néanmoins, la réflexion engagée au sein du RMT (Tableau 3) s'est révélée utile sur deux points : d'une part la typologie des utilisations possibles, d'autre part le fait que les études de cas qui ont pu être menées couvrent différentes dimensions de ces utilisations possibles, preuve de la richesse potentielle de l'approche.
Le couplage avec d'autres sources de données non biologiques (sol, RGA, climat) et le recours à de nouvelles démarches statistiques et de nouveaux outils d'évaluation suscitent l'intérêt. Le partage des données en santé végétale devrait donc pouvoir profiter pleinement des développements récents en termes de méthodes de calcul et d'exploitation de données massives.
Pour dépasser la simple analyse de faisabilité effectuée jusqu'ici, il est désormais indispensable de co-construire un ou des projets de recherche pour disposer des moyens matériels et humains nécessaires au traitement concret de certains des cas abordés. Ceci permettrait de valider les preuves de concepts, et d'envisager des actions en collaboration avec les détenteurs de données, comme c'est déjà le cas pour certains pathosystèmes tels que celui du mildiou de la pomme de terre, ou envisageable pour d'autres comme Ditylenchus.
Le partage des données en santé végétale est un enjeu important qu'il convient de traiter pour mutualiser des moyens afin de répondre au besoin en connaissances lié à l'augmentation de la fréquence des émergences ou au développement du biocontrôle. Il profitera certainement des progrès récents en termes de méthodes de calcul, de la bio-informatique, ou du séquençage haut débit...
POUR EN SAVOIR PLUS
CONTACTS : didier.andrivon@inra.fr
andre.chabert@acta.asso.fr
BIBLIOGRAPHIE : - A Buisson, A Chabert, L Ruck, S Fournet, Nematodes associated with damage in oilseed rape: new data on the biology and geographical distribution of Meloidogyne artiellia, Nematology 2014 16 (2), 201-206.
- L. Vinsant-Le Lous et J. Janson, Enquête en culture sur les nématodes Ditylenchus dipsaci et Ditylenchus gigas sur féverole, Rapport de synthèse FNAMS 2011, 2012 et 2013.
 Imprimer
Imprimer